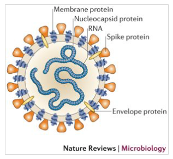
Covid 19 - État de l'art
L’émergence d'un nouveau coronavirus, SARS-CoV-2, en Chine, en décembre 2019, est à l’origine d’une épidémie mondiale, source d’inquiétudes et de questionnements, souvent justifiés mais parfois aussi démesurés. En cette période de confinement, nous sommes inondés d'informations et de prises de paroles, qui peuvent se révéler anxiogènes, inexactes ou encore volontairement polémiques. Cependant, de nombreuses études scientifiques sont publiées quotidiennement et souvent en accès libre : elles peuvent nous permettre de faire la part des choses et de mieux saisir les enjeux de cette épidémie de COVID-19.
EspritsCo vous propose un modeste état de l'art des travaux et études menés par la communauté scientifique mondiale avec un premier volet consacré aux principales questions concernant SARS-CoV-2 et la maladie qu'il induit COVID-19.
Pour aller plus loin
1. Les sites d’information de référence
- SRLF - Société de réanimation de langue Française
- INSERM - Dossier d’information Coronavirus et COVID-19
- Société Française de pharmacologie et de thérapeutique
- Santé Publique France
- Infovac-France : la plateforme d’information sur la vaccination
- Direction Générale de la Santé,
- OMS – Organisation Mondiale de la santé
- CDC – Center for disease control and prevention
- Institut John Hopkins
2. Suivre l’évolution de la pandémie dans le monde
- Infographie de la cinétique de propagation de COVID-19 dans le monde et en France par santé publique France.
- Suivi quotidien de l’évolution de l’épidémie dans le monde à partir des chiffres consolidés de l’université Johns Hopkins.
- Covid-19 Dashboard par Pratap Vardhan fondé sur les chiffres de Johns Hopkins.
3. Vulgarisation scientifique
Références bibliographiques
1. Chen Y, Liu Q, Guo D. Emerging coronaviruses: Genome structure, replication, and pathogenesis. J Med Virol. 2020;92(4):418-423. doi:10.1002/jmv.25681
2. Chan JF-W, To KK-W, Tse H, Jin D-Y, Yuen K-Y. Interspecies transmission and emergence of novel viruses: lessons from bats and birds. Trends Microbiol. 2013;21(10):544-555. doi:10.1016/j.tim.2013.05.005
3. Chan JF-W, Kok K-H, Zhu Z, et al. Genomic characterization of the 2019 novel human-pathogenic coronavirus isolated from a patient with atypical pneumonia after visiting Wuhan. Emerg Microbes Infect. 2020;9(1):221-236. doi:10.1080/22221751.2020.1719902
4. Zhou P, Yang X-L, Wang X-G, et al. A pneumonia outbreak associated with a new coronavirus of probable bat origin. Nature. 2020;579(7798):270-273. doi:10.1038/s41586-020-2012-7
5. Fan Y, Zhao K, Shi Z-L, Zhou P. Bat Coronaviruses in China. Viruses. 2019;11(3):210. doi:10.3390/v11030210
6. Hu B, Zeng L-P, Yang X-L, et al. Discovery of a rich gene pool of bat SARS-related coronaviruses provides new insights into the origin of SARS coronavirus. Drosten C, ed. PLOS Pathog. 2017;13(11):e1006698. doi:10.1371/journal.ppat.1006698
7. Guan Y. Isolation and Characterization of Viruses Related to the SARS Coronavirus from Animals in Southern China. Science. 2003;302(5643):276-278. doi:10.1126/science.1087139
8. Lau SKP, Woo PCY, Li KSM, et al. Severe acute respiratory syndrome coronavirus-like virus in Chinese horseshoe bats. Proc Natl Acad Sci. 2005;102(39):14040-14045. doi:10.1073/pnas.0506735102
9. Song H-D, Tu C-C, Zhang G-W, et al. Cross-host evolution of severe acute respiratory syndrome coronavirus in palm civet and human. Proc Natl Acad Sci. 2005;102(7):2430-2435. doi:10.1073/pnas.0409608102
10. Graham RL, Donaldson EF, Baric RS. A decade after SARS: strategies for controlling emerging coronaviruses. Nat Rev Microbiol. 2013;11(12):836-848. doi:10.1038/nrmicro3143
11. Tu C, Crameri G, Kong X, et al. Antibodies to SARS Coronavirus in Civets. Emerg Infect Dis. 2004;10(12):2244-2248. doi:10.3201/eid1012.040520
12. The Chinese SARS Molecular Epidemiology Consortium. Molecular Evolution of the SARS Coronavirus During the Course of the SARS Epidemic in China. Science. 2004;303(5664):1666-1669. doi:10.1126/science.1092002
13. Yang Z -y., Werner HC, Kong W -p., et al. Evasion of antibody neutralization in emerging severe acute respiratory syndrome coronaviruses. Proc Natl Acad Sci. 2005;102(3):797-801. doi:10.1073/pnas.0409065102
14. Bolles M, Donaldson E, Baric R. SARS-CoV and emergent coronaviruses: viral determinants of interspecies transmission. Curr Opin Virol. 2011;1(6):624-634. doi:10.1016/j.coviro.2011.10.012
15. Graham RL, Baric RS. Recombination, Reservoirs, and the Modular Spike: Mechanisms of Coronavirus Cross-Species Transmission. J Virol. 2010;84(7):3134-3146. doi:10.1128/JVI.01394-09
16. Zhang T, Wu Q, Zhang Z. Probable Pangolin Origin of SARS-CoV-2 Associated with the COVID-19 Outbreak. Curr Biol. 2020;30(7):1346-1351.e2. doi:10.1016/j.cub.2020.03.022
17. Wong MC, Javornik Cregeen SJ, Ajami NJ, Petrosino JF. Evidence of Recombination in Coronaviruses Implicating Pangolin Origins of NCoV-2019. Microbiology; 2020. doi:10.1101/2020.02.07.939207
18. Xiao K, Zhai J, Feng Y, et al. Isolation and Characterization of 2019-NCoV-like Coronavirus from Malayan Pangolins. Microbiology; 2020. doi:10.1101/2020.02.17.951335
19. Chu DKW, Pan Y, Cheng SMS, et al. Molecular Diagnosis of a Novel Coronavirus (2019-nCoV) Causing an Outbreak of Pneumonia. Clin Chem. 2020;66(4):549-555. doi:10.1093/clinchem/hvaa029
20. Corman VM, Landt O, Kaiser M, et al. Detection of 2019 novel coronavirus (2019-nCoV) by real-time RT-PCR. Eurosurveillance. 2020;25(3). doi:10.2807/1560-7917.ES.2020.25.3.2000045
21. Zou L, Ruan F, Huang M, et al. SARS-CoV-2 Viral Load in Upper Respiratory Specimens of Infected Patients. N Engl J Med. 2020;382(12):1177-1179. doi:10.1056/NEJMc2001737
22. Wu Y, Guo C, Tang L, et al. Prolonged presence of SARS-CoV-2 viral RNA in faecal samples. Lancet Gastroenterol Hepatol. 2020;5(5):434-435. doi:10.1016/S2468-1253(20)30083-2
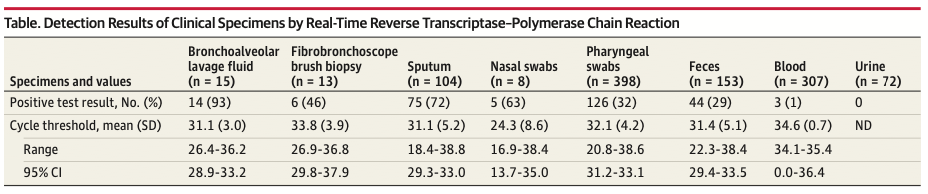
23. Wang W, Xu Y, Gao R, et al. Detection of SARS-CoV-2 in Different Types of Clinical Specimens. JAMA. March 2020. doi:10.1001/jama.2020.3786
24. Liu Y, Yan L-M, Wan L, et al. Viral dynamics in mild and severe cases of COVID-19. Lancet Infect Dis. March 2020:S1473309920302322. doi:10.1016/S1473-3099(20)30232-2
25. Lescure F-X, Bouadma L, Nguyen D, et al. Clinical and virological data of the first cases of COVID-19 in Europe: a case series. Lancet Infect Dis. March 2020:S1473309920302000. doi:10.1016/S1473-3099(20)30200-0
26. Chen L, Lou J, Bai Y, Wang M. COVID-19 Disease With Positive Fecal and Negative Pharyngeal and Sputum Viral Tests: Am J Gastroenterol. March 2020:1. doi:10.14309/ajg.0000000000000610
27. Guo L, Ren L, Yang S, et al. Profiling Early Humoral Response to Diagnose Novel Coronavirus Disease (COVID-19). Clin Infect Dis. March 2020:ciaa310. doi:10.1093/cid/ciaa310
28. Biggerstaff M, Cauchemez S, Reed C, Gambhir M, Finelli L. Estimates of the reproduction number for seasonal, pandemic, and zoonotic influenza: a systematic review of the literature. BMC Infect Dis. 2014;14(1):480. doi:10.1186/1471-2334-14-480
29. Liu Y, Gayle AA, Wilder-Smith A, Rocklöv J. The reproductive number of COVID-19 is higher compared to SARS coronavirus. J Travel Med. 2020;27(2):taaa021. doi:10.1093/jtm/taaa021
30. Choi S, Jung E, Choi BY, Hur YJ, Ki M. High reproduction number of Middle East respiratory syndrome coronavirus in nosocomial outbreaks: mathematical modelling in Saudi Arabia and South Korea. J Hosp Infect. 2018;99(2):162-168. doi:10.1016/j.jhin.2017.09.017
31. Wu JT, Leung K, Leung GM. Nowcasting and forecasting the potential domestic and international spread of the 2019-nCoV outbreak originating in Wuhan, China: a modelling study. The Lancet. 2020;395(10225):689-697. doi:10.1016/S0140-6736(20)30260-9
32. Rothe C, Schunk M, Sothmann P, et al. Transmission of 2019-nCoV Infection from an Asymptomatic Contact in Germany. N Engl J Med. 2020;382(10):970-971. doi:10.1056/NEJMc2001468
33. Ganyani T, Kremer C, Chen D, et al. Estimating the Generation Interval for COVID-19 Based on Symptom Onset Data. Infectious Diseases (except HIV/AIDS); 2020. doi:10.1101/2020.03.05.20031815
34. Nishiura H, Linton NM, Akhmetzhanov AR. Serial interval of novel coronavirus (COVID-19) infections. Int J Infect Dis. 2020;93:284-286. doi:10.1016/j.ijid.2020.02.060
35. Li Q, Guan X, Wu P, et al. Early Transmission Dynamics in Wuhan, China, of Novel Coronavirus–Infected Pneumonia. N Engl J Med. 2020;382(13):1199-1207. doi:10.1056/NEJMoa2001316
36. Yu P, Zhu J, Zhang Z, Han Y. A Familial Cluster of Infection Associated With the 2019 Novel Coronavirus Indicating Possible Person-to-Person Transmission During the Incubation Period. J Infect Dis. February 2020:jiaa077. doi:10.1093/infdis/jiaa077
37. Bai Y, Yao L, Wei T, et al. Presumed Asymptomatic Carrier Transmission of COVID-19. JAMA. 2020;323(14):1406. doi:10.1001/jama.2020.2565
38. Li R, Pei S, Chen B, et al. Substantial undocumented infection facilitates the rapid dissemination of novel coronavirus (SARS-CoV2). Science. March 2020:eabb3221. doi:10.1126/science.abb3221
39. Han Y, Yang H. The transmission and diagnosis of 2019 novel coronavirus infection disease (COVID‐19): A Chinese perspective. J Med Virol. March 2020:jmv.25749. doi:10.1002/jmv.25749
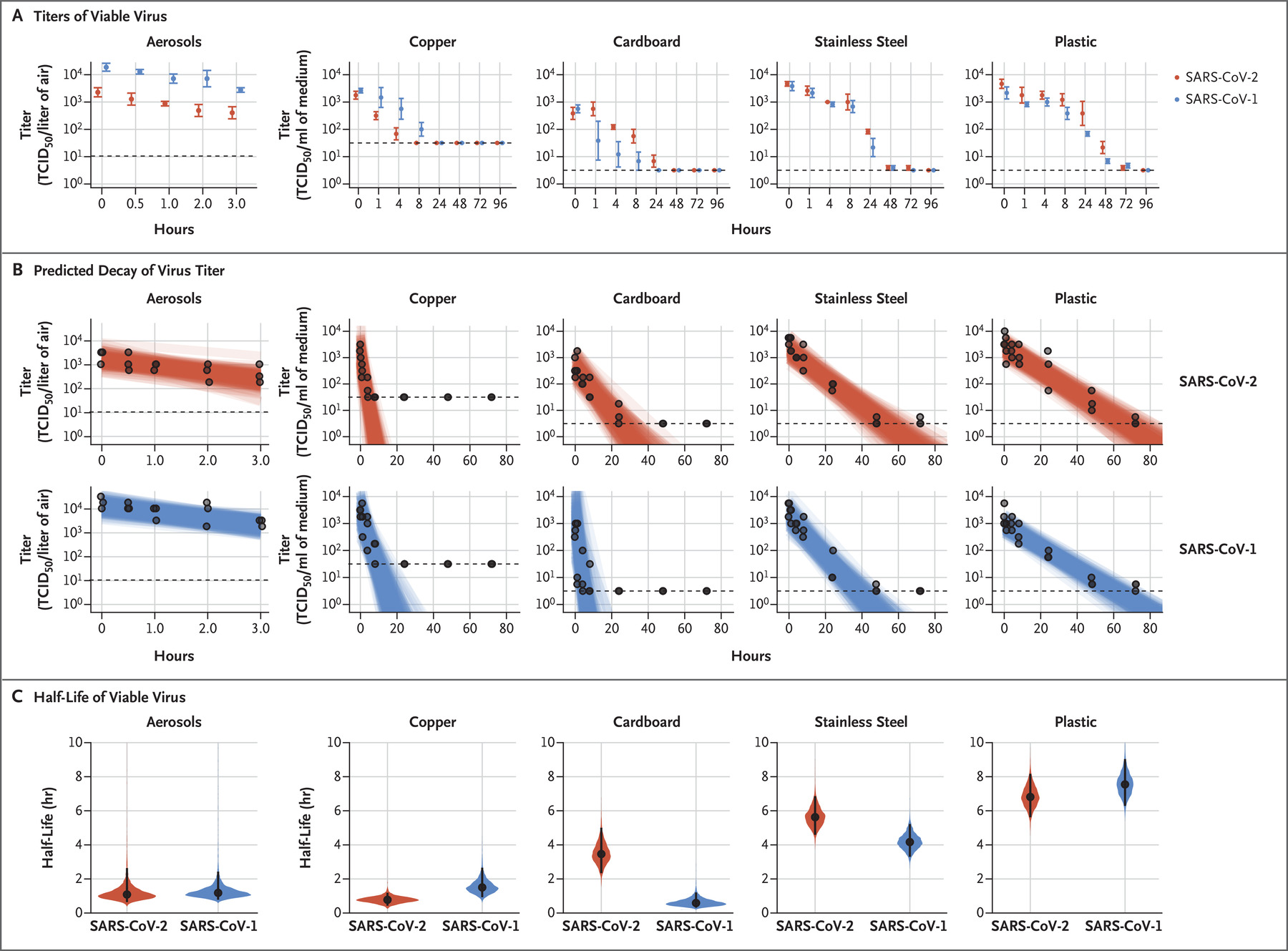
40. van Doremalen N, Bushmaker T, Morris DH, et al. Aerosol and Surface Stability of SARS-CoV-2 as Compared with SARS-CoV-1. N Engl J Med. March 2020:NEJMc2004973. doi:10.1056/NEJMc2004973
41. Wang D, Hu B, Hu C, et al. Clinical Characteristics of 138 Hospitalized Patients With 2019 Novel Coronavirus–Infected Pneumonia in Wuhan, China. JAMA. 2020;323(11):1061. doi:10.1001/jama.2020.1585
42. Han R, Huang L, Jiang H, Dong J, Peng H, Zhang D. Early Clinical and CT Manifestations of Coronavirus Disease 2019 (COVID-19) Pneumonia. Am J Roentgenol. March 2020:1-6. doi:10.2214/AJR.20.22961
43. Gu J, Han B, Wang J. COVID-19: Gastrointestinal Manifestations and Potential Fecal–Oral Transmission. Gastroenterology. March 2020:S001650852030281X. doi:10.1053/j.gastro.2020.02.054
44. Brann DH, Tsukahara T, Weinreb C, et al. Non-Neuronal Expression of SARS-CoV-2 Entry Genes in the Olfactory System Suggests Mechanisms Underlying COVID-19-Associated Anosmia. Neuroscience; 2020. doi:10.1101/2020.03.25.009084
45. Iacobucci G. Sixty seconds on . . . anosmia. BMJ. March 2020:m1202. doi:10.1136/bmj.m1202
46. Eliezer M, Hautefort C, Hamel A-L, et al. Sudden and Complete Olfactory Loss Function as a Possible Symptom of COVID-19. JAMA Otolaryngol Neck Surg. April 2020. doi:10.1001/jamaoto.2020.0832
47. Gautier J-F, Ravussin Y. A New Symptom of COVID-19: Loss of Taste and Smell. Obesity. April 2020. doi:10.1002/oby.22809
48. Vaira LA, Salzano G, Deiana G, De Riu G. Anosmia and Ageusia: Common Findings in COVID-19 Patients: Otolaryngological Manifestations in COVID-19. The Laryngoscope. April 2020. doi:10.1002/lary.28692
49. Recalcati S. Cutaneous manifestations in COVID-19: a first perspective. J Eur Acad Dermatol Venereol. March 2020. doi:10.1111/jdv.16387
50. Wu P, Duan F, Luo C, et al. Characteristics of Ocular Findings of Patients With Coronavirus Disease 2019 (COVID-19) in Hubei Province, China. JAMA Ophthalmol. March 2020. doi:10.1001/jamaophthalmol.2020.1291
51. Wu Z, McGoogan JM. Characteristics of and Important Lessons From the Coronavirus Disease 2019 (COVID-19) Outbreak in China: Summary of a Report of 72 314 Cases From the Chinese Center for Disease Control and Prevention. JAMA. 2020;323(13):1239. doi:10.1001/jama.2020.2648
52. Zhou F, Yu T, Du R, et al. Clinical course and risk factors for mortality of adult inpatients with COVID-19 in Wuhan, China: a retrospective cohort study. The Lancet. 2020;395(10229):1054-1062. doi:10.1016/S0140-6736(20)30566-3
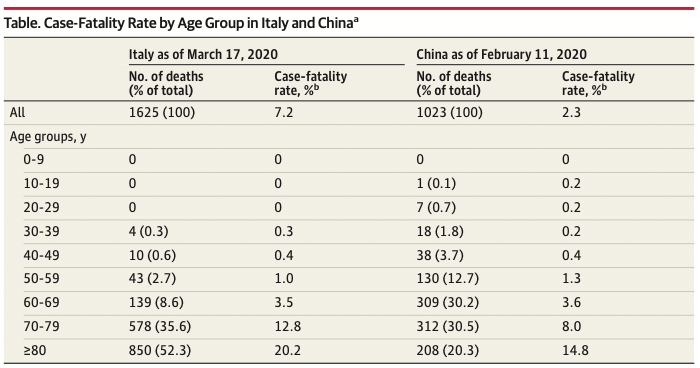
53. Onder G, Rezza G, Brusaferro S. Case-Fatality Rate and Characteristics of Patients Dying in Relation to COVID-19 in Italy. JAMA. March 2020. doi:10.1001/jama.2020.4683
54. Baud D, Qi X, Nielsen-Saines K, Musso D, Pomar L, Favre G. Real estimates of mortality following COVID-19 infection. Lancet Infect Dis. March 2020:S147330992030195X. doi:10.1016/S1473-3099(20)30195-X
55. Ji Y, Ma Z, Peppelenbosch MP, Pan Q. Potential association between COVID-19 mortality and health-care resource availability. Lancet Glob Health. 2020;8(4):e480. doi:10.1016/S2214-109X(20)30068-1